有用的dump
1. 如何由lammps的轨迹转化为POSCAR
构建机器学习势时,通常需要主动学习。主动学习就需要将用MLIAP跑出来的结果返回到DFT中进行计算。
具体来讲,要将分子动力学轨迹转化为第一性原理的输入格式。(如从lammps到vasp)
lammps并不直接支持输出POSCAR格式,比较容易想到的思路是让lammps输出xyz文件,再有xyz文件转化为POSCAR。
不过麻烦的一点是,lammps输出xyz文件并不带有晶格大小的信息,无法转化为POSCAR。
这里,我目前的方案是,先用custom style的dump输出文件,如下:
dump 1 all custom 1 dump20nvt id element x y z
dump_modify 1 sort id element O Zr Y H
为了避免输出lammps中的type,而不是元素符号,这里修饰一下dump(即 element O Zr Y H),这里O、Zr、Y、H分别对应 type 1、2、3、4
sort id 必不可少,保证原子对应。
这样得到的dump文件格式,如下:
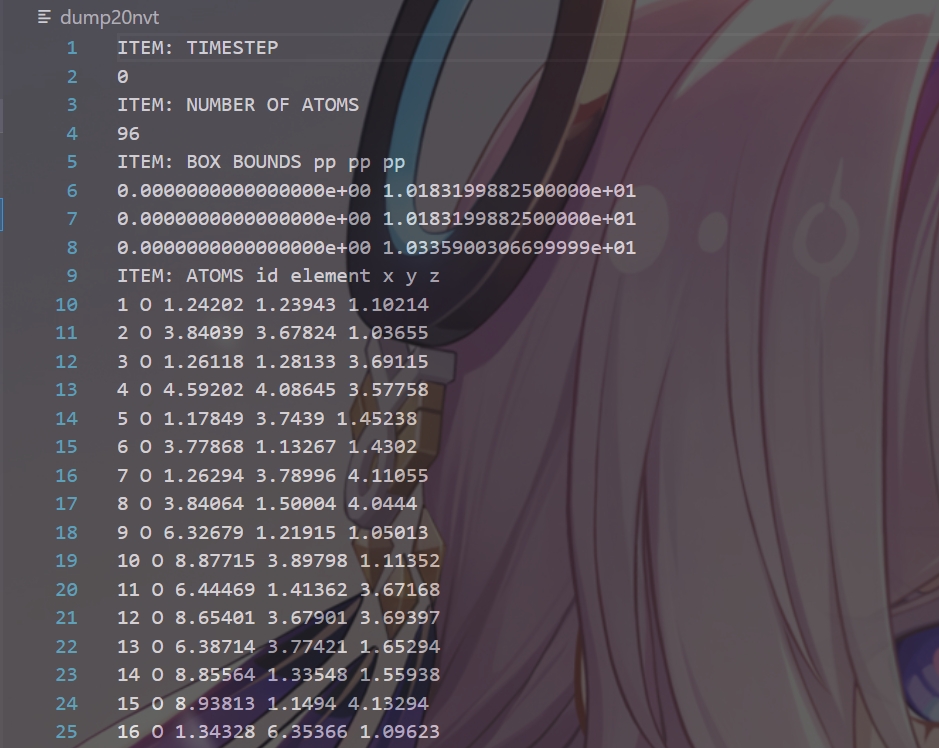
可以看到,同时包含了元素符号、晶格和原子坐标信息。其实这个自定义风格,custom,就是稍加修改的atom风格。
剩下的问题就是,如何将lammps的dump文件转化为xyz文件,再由xyz文件转化为POSCAR。
OVITO模块读取dump文件比较好用,个人觉得比ase的强,所以这里采用ovito模块,如下:
from ovito.io import import_file,export_file
pipeline = import_file('dump')
for frame,data in enumerate(pipeline.frames):
if frame < 10:
export_file(data,"xyz{}".format(frame),format="xyz",columns =["Particle Identifier", "Particle Type", "Position.X", "Position.Y", "Position.Z"])
这里简单测试一下,只输出了前10个结构。结果很成功。
然后就是把xyz文件转化为POSCAR,这里用ase模块就行,如下:
from ase.io import read,write
for i in range(10):
at = read("xyz{}".format(i))
write('POSCAR{}'.format(i),at,format='vasp')